인체의 비밀 풀어낼 사상 최대 규모 '단백질 지도' 완성
-
기사 스크랩
-
공유
-
댓글
-
클린뷰
-
프린트
유전자 부호화 단백질 8천200여종, 상호작용 5만2천500여개 망라
미국·캐나다 등 공동 연구진, 저널 '네이처'에 논문
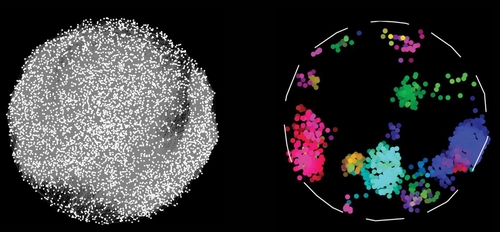
인체를 구성하는 세포 하나하나는, 유전자와 단백질 등 구성 물질의 무수한 상호작용을 통해 생성되고 유지된다.
우리 몸에는 약 2만 개의 단백질 부호화 유전자(protein-coding gene)가 있다.
그런데 이들 유전자의 암호가 풀려 생성되는 단백질의 상호작용 가운데 어떤 부분이 건강에 이롭고, 어떤 부분이 질병을 일으키는지는 대부분 밝혀지지 않았다.
그래서 미국 다나-파버 암 연구소와 캐나다 토론토대 과학자들은 1990년대 중반부터 이른바 '상호작용체 지도(interactome map)'를 만드는 장기 연구 프로젝트를 추진해 왔다.
유전자 암호의 해독으로 생성되는 단백질의 상호작용을 일목요연하게 보여주는 '인간 단백질 지도'를 만들자는 취지였다.
이 프로젝트엔 미국과 캐나다 외에 스페인, 벨기에, 프랑스, 이스라엘 등 6개국 과학자 80여 명이 참여했다.
마침내 이 다국적 공동 연구가 열매를 맺어 사상 최대 규모의 '인간 단백질 지도'가 완성됐다.
관련 논문은 저널 '네이처(Nature)'에 실렸다.
'인간 참고 상호작용체(HuRI)'로 명명된 이 지도에는 인간 단백질 8천275종과 이들 단백질이 일으키는 5만2천569개 유형의 상호작용이 기록됐다.
다나-파버 암 연구소와 토론토대는 작년에도 작은 버전의 단백질 지도를 만들었다.
하지만 그때는 단백질 상호작용 유형이 1만4천 개로 약 4분의 1에 불과했다.

9일 온라인(www.eurekalert.org)에 공개된 논문 개요에 따르면 이 단백질 지도는 향후 연구에 발판 역할을 할 '단백질 정보의 비계(scaffold of information)'와 같다.
고장 난 유전자가 어떻게 암과 같은 질병을 일으키는지, 신종 코로나바이러스 같은 바이러스가 어떻게 인간의 단백질과 상호작용하는지 등을 더 잘 이해하게 돕는 실질적인 정보가 담겨 있다.
토론토대 세포 생물분자 연구소의 프레데릭 로스 교수는 "이 지도를 활용하면 어떤 단백질의 기능을 예측하는 것도 가능하다"라면서 "함께 작용하는 단백질을 보면 실마리를 찾을 수 있다"라고 말했다.
실제로 HuRI 지도는 이미, 인간 단백질과 관련된 세포의 새로운 역할, 병세를 악화하는 분자 수준의 문제, 세포 예정사에 관여하는 단백질의 새로운 기능 등을 밝혀내는 데 요긴히 활용된 것으로 전해진다.
과학자들은 이번 연구에서 '연좌(guilt by association)' 원칙을 적용했다.
두 종의 단백질이 각각 상호작용하는 단백질이 유사하면, 비슷한 생리 과정에 관여하는 것으로 보는 것이다.
연구팀은 단백질 1만7천500종에서 나올 수 있는 모든 2개 단백질 조합(pairwise combinations)에 대해 일일이 상호작용 능력을 테스트했다.
효모균에 발현시킨 각 조합의 두 단백질이 상호작용을 일으키면, 분자 스위치처럼 작용해 효모균의 성장을 촉진했다.
조합마다 각각 3종의 효모균에 테스트해, 전체 테스트는 무려 30억 회에 달했지만. 이 가운데 신뢰도가 높은 것들만 골라내 논문에 인용했다.
이번에 공개된 단백질 지도는 사상 최대인데도 체내에서 벌어지는 단백질 상호작용의 2% 내지 11%만 반영된 것이라고 한다.
효모균에는 단백질 기능에 필요한 인간 특유의 분자 인자가 없다는 점 등이 실험의 한계로 작용했다.
그러나 미국 '콜드 스프링 하버 연구소'의 바이오 아카이브(www.bioRxiv)에 업로드된 예전 버전의 HuRI 지도는 반응이 뜨거웠다.
2019년 4월 첫 공개 이후 1만5천여 명이 전체 데이터 세트를 다운로드했다.
로스 교수는 "규모가 3분의 1에도 못 미치는 이전의 연구가 800회 이상 다른 논문에 인용됐다"라면서 "이번 연구 결과도 크게 주목받을 거로 기대한다"라고 말했다.
/연합뉴스
미국·캐나다 등 공동 연구진, 저널 '네이처'에 논문

우리 몸에는 약 2만 개의 단백질 부호화 유전자(protein-coding gene)가 있다.
그런데 이들 유전자의 암호가 풀려 생성되는 단백질의 상호작용 가운데 어떤 부분이 건강에 이롭고, 어떤 부분이 질병을 일으키는지는 대부분 밝혀지지 않았다.
그래서 미국 다나-파버 암 연구소와 캐나다 토론토대 과학자들은 1990년대 중반부터 이른바 '상호작용체 지도(interactome map)'를 만드는 장기 연구 프로젝트를 추진해 왔다.
유전자 암호의 해독으로 생성되는 단백질의 상호작용을 일목요연하게 보여주는 '인간 단백질 지도'를 만들자는 취지였다.
이 프로젝트엔 미국과 캐나다 외에 스페인, 벨기에, 프랑스, 이스라엘 등 6개국 과학자 80여 명이 참여했다.
마침내 이 다국적 공동 연구가 열매를 맺어 사상 최대 규모의 '인간 단백질 지도'가 완성됐다.
관련 논문은 저널 '네이처(Nature)'에 실렸다.
'인간 참고 상호작용체(HuRI)'로 명명된 이 지도에는 인간 단백질 8천275종과 이들 단백질이 일으키는 5만2천569개 유형의 상호작용이 기록됐다.
다나-파버 암 연구소와 토론토대는 작년에도 작은 버전의 단백질 지도를 만들었다.
하지만 그때는 단백질 상호작용 유형이 1만4천 개로 약 4분의 1에 불과했다.

9일 온라인(www.eurekalert.org)에 공개된 논문 개요에 따르면 이 단백질 지도는 향후 연구에 발판 역할을 할 '단백질 정보의 비계(scaffold of information)'와 같다.
고장 난 유전자가 어떻게 암과 같은 질병을 일으키는지, 신종 코로나바이러스 같은 바이러스가 어떻게 인간의 단백질과 상호작용하는지 등을 더 잘 이해하게 돕는 실질적인 정보가 담겨 있다.
토론토대 세포 생물분자 연구소의 프레데릭 로스 교수는 "이 지도를 활용하면 어떤 단백질의 기능을 예측하는 것도 가능하다"라면서 "함께 작용하는 단백질을 보면 실마리를 찾을 수 있다"라고 말했다.
실제로 HuRI 지도는 이미, 인간 단백질과 관련된 세포의 새로운 역할, 병세를 악화하는 분자 수준의 문제, 세포 예정사에 관여하는 단백질의 새로운 기능 등을 밝혀내는 데 요긴히 활용된 것으로 전해진다.
과학자들은 이번 연구에서 '연좌(guilt by association)' 원칙을 적용했다.
두 종의 단백질이 각각 상호작용하는 단백질이 유사하면, 비슷한 생리 과정에 관여하는 것으로 보는 것이다.
연구팀은 단백질 1만7천500종에서 나올 수 있는 모든 2개 단백질 조합(pairwise combinations)에 대해 일일이 상호작용 능력을 테스트했다.
효모균에 발현시킨 각 조합의 두 단백질이 상호작용을 일으키면, 분자 스위치처럼 작용해 효모균의 성장을 촉진했다.
조합마다 각각 3종의 효모균에 테스트해, 전체 테스트는 무려 30억 회에 달했지만. 이 가운데 신뢰도가 높은 것들만 골라내 논문에 인용했다.
이번에 공개된 단백질 지도는 사상 최대인데도 체내에서 벌어지는 단백질 상호작용의 2% 내지 11%만 반영된 것이라고 한다.
효모균에는 단백질 기능에 필요한 인간 특유의 분자 인자가 없다는 점 등이 실험의 한계로 작용했다.
그러나 미국 '콜드 스프링 하버 연구소'의 바이오 아카이브(www.bioRxiv)에 업로드된 예전 버전의 HuRI 지도는 반응이 뜨거웠다.
2019년 4월 첫 공개 이후 1만5천여 명이 전체 데이터 세트를 다운로드했다.
로스 교수는 "규모가 3분의 1에도 못 미치는 이전의 연구가 800회 이상 다른 논문에 인용됐다"라면서 "이번 연구 결과도 크게 주목받을 거로 기대한다"라고 말했다.
/연합뉴스



![K팝 업계에도 '친환경' 바람…폐기물 되는 앨범은 '골칫거리' [연계소문]](https://img.hankyung.com/photo/202206/99.27464274.3.jpg)


